Cluster():层次聚类的第一步
当你用 get_distance_matrix() 得到一个已经计算好的的距离矩阵(n×n 方形的矩阵)后,就可以开始做聚类了。这是序列分析里很常见的一步。
层次聚类(hierarchical clustering)是一种无监督机器学习算法(unsupervised machine learning algorithm),简单直接、上手容易,能帮你看出数据中典型的轨迹模式。
层次聚类的常见流程
我们可以用
get_distance_matrix()准备一个距离矩阵D(形状n×n),同时准备一份实体 ID 列表/数组entity_ids(长度为n,全部非重复)。我们可以创建一个
Cluster()实例,必要时你也可以画一张树状图(dendrogram)。我们可以选定簇数
k(clusters 的数量),拿到聚类标签。我们可以用
ClusterQuality()对不同的k做比较,查看聚类质量指标(cluster quality indicators,CQI)。我们可以用
ClusterResults()导出成员表(cluster membership table,每个实体分别属于哪个簇),并绘制各簇占比图。
我们做完上述步骤后,接下来可以常见的下游分析:
1)用状态分布图或索引图分别可视化各个簇;
2)在回归模型里使用每个个体的簇标签,既可以作为自变量,也可以作为因变量。
下面是把 Cluster()、ClusterQuality() 和 ClusterResults() 组合起来做层次聚类的一个示例。
from sequenzo.clustering.hierarchical_clustering import Cluster, ClusterQuality, ClusterResults
# ---------------------
# distance_matrix: 距离矩阵 (n x n)
# ids: 长度为 n 的非重复 ID 列表/数组
# 一般推荐 "ward"
cluster = Cluster(
matrix=distance_matrix,
entity_ids=ids,
clustering_method="ward"
)
# 可选:画树状图(n 很大时会自动隐藏标签)
# 新手建议先试一下,帮助你直观看到层次结构
cluster.plot_dendrogram(
# 不想保存就删掉这个参数
save_as="dendrogram.png",
# 图标题
title="Dendrogram"
)
# ---------------------
# 用聚类质量指标(CQI)来帮助选择 k
cluster_quality = ClusterQuality(cluster, max_clusters=20)
cluster_quality.compute_cluster_quality_scores()
# 打印CQI表格信息,包含着每个CQI指标下计算得出的最优簇数 k 是什么
print(cluster_quality.get_cqi_table())
# 想改保存文件名也可以
cluster_quality.plot_cqi_scores(save_as="quality.png")
# ---------------------
# 导出结果
cluster_results = ClusterResults(cluster)
# 假设 CQI 图建议 k=6
# 取出簇成员表,表中包含两列:实体 ID 与 Cluster
members = cluster_results.get_cluster_memberships(num_clusters=6)
# 查看每个簇的样本数与占比
distribution = cluster_results.get_cluster_distribution(num_clusters=6)
cluster_results.plot_cluster_distribution(num_clusters=6,
save_as="cluster_sizes.png")接下来,我们在这个函数文档里重点介绍 Cluster()。在实现层面,它使用了 Python 包 fastcluster,相较于 SciPy 的标准做法,它通常更快、更省内存,对大数据集尤其友好。
Cluster() 的用法
from sequenzo.clustering.hierarchical_clustering import Cluster
cluster = Cluster(
matrix=distance_matrix, # n×n 的对称方形矩阵
entity_ids=ids, # 长度为 n(n 是数据里的实体数,也就是样本数),全部非重复
clustering_method="ward" # 可以选择"ward" | "single" | "complete" | "average" | "centroid" | "median"
)参数(Entry Parameters)
| 参数 | 必填 | 类型 | 说明 |
|---|---|---|---|
matrix | ✓ | array/DataFrame | 已经计算好的完整距离矩阵,形状 n×n。 |
entity_ids | ✓ | array-like | 长度为 n 的非重复 ID 列表/数组,顺序需与 matrix 的行/列一致。 |
clustering_method | ✗ | str | 链接方法(linkage method)。默认 "ward"。可选 "single"、"complete"、"average"、"centroid"、"median"。 |
这个函数做了什么
校验输入:检查是否为矩阵、
entity_ids的长度是否匹配、ID 是否非重复。处理矩阵里的“小毛病”:
如果有
NaN/Inf,用矩阵里最大的有限值进行替换。在容差范围内,做对称化:
(D + D.T) / 2。
把方阵转换成压缩形式(condensed),再用高效的层次聚类程序计算链接矩阵(linkage matrix)。
保存链接矩阵,便于后续画树状图(dendrograms)或剪树(cut the tree)。
返回对象(Returned Object)
返回一个 Cluster 实例,主要属性:
entity_ids:你的 ID(numpy 数组,长度n)。full_matrix:校验过的 n×n 距离矩阵(numpy 数组)。condensed_matrix:供链接算法使用的压缩向量。linkage_matrix:层次聚类得到的链接矩阵。
主要算法:
plot_dendrogram(...):渲染或保存树状图。get_cluster_labels(num_clusters):返回长度为n的簇标签数组。
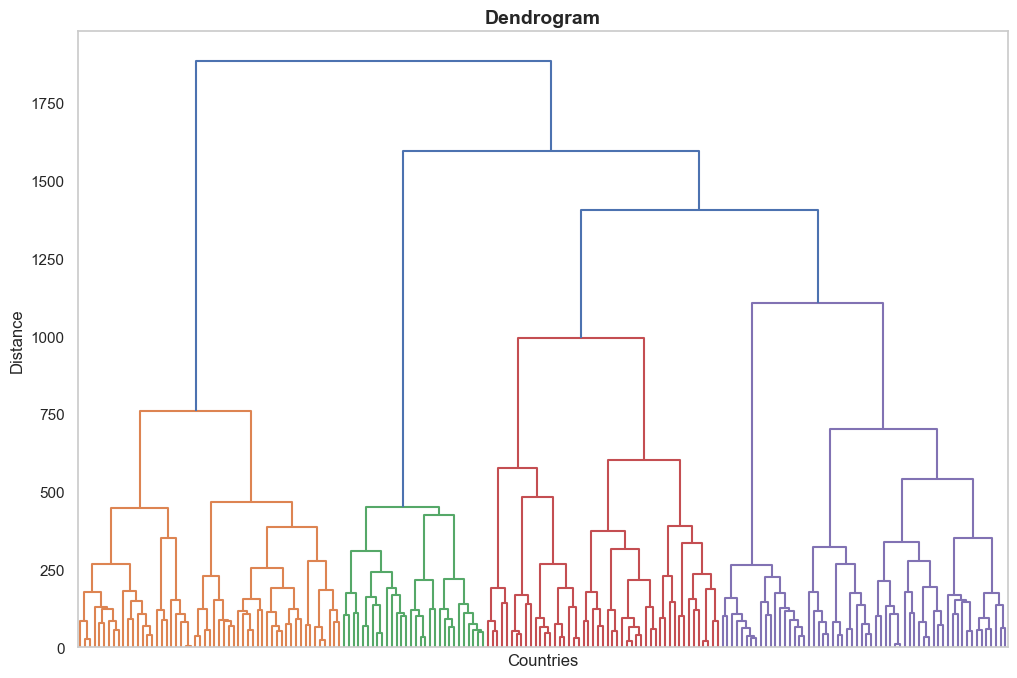
函数方法一:plot_dendrogram()
渲染或保存已拟合层次聚类的树状图。
用法
cluster.plot_dendrogram(
save_as=None, # 例如 "dendrogram.png";为 None 则只显示,不保存
style="whitegrid", # seaborn 的风格
title="Dendrogram",
xlabel="Entities", # 可以根据你的数据属性来更改名字,比如"Individuals","Countries"
ylabel="Distance",
grid=False,
dpi=200,
figsize=(12, 8)
)参数(Entry parameters)
| 参数 | 必填 | 类型 | 说明 |
|---|---|---|---|
save_as | ✗ | str 或 None | 图像保存路径(如 "dendrogram.png")。为 None 则只显示不保存。 |
style | ✗ | str | seaborn 风格名。常用有 "whitegrid"、"darkgrid"、"white"、"dark"、"ticks"。 |
title | ✗ | str | 图标题,默认 "Dendrogram"。 |
xlabel | ✗ | str | X 轴标题,默认 "Entities"。 |
ylabel | ✗ | str | Y 轴标题,默认 "Distance"。 |
grid | ✗ | bool | 是否显示网格线,默认 False。 |
dpi | ✗ | int | 保存图像时的分辨率,默认 200。 |
figsize | ✗ | tuple(float, float) | 图像尺寸(英寸),默认 (12, 8)。 |
关于
style与grid:
style(如"whitegrid"、"darkgrid")会设定整体主题,可能包含背景、字体、网格线等。grid是显式覆盖项:grid=False会关闭网格线,即使所选风格默认带网格。grid的开关优先于风格的默认网格设置。在我们实际使用中,你可以多尝试几个组合,选你喜欢的就可以。
这个Cluster()做了什么
用已有的
linkage_matrix绘制树状图。当数据的样本量 n 很大时,叶子标签会被隐藏,避免图面拥挤。如果需要小规模标注,建议用
scipy.cluster.hierarchy.dendrogram自行绘制。如果你提供了
save_as这个参数,则意味着你想要保存图片,函数会根据另一个参数dpi看保存到图片高清度需要多少(默认 200);如果没传save_as,这张图默认会显示出来,不会保存。
返回值
- 无(显示和/或保存图片)。
注意:如果你的距离矩阵曾含有 NaN/Inf 或轻微不对称,Cluster() 在计算链接前会进行修复,并可能给出警告。但这不是错误提示。
例子
cluster.plot_dendrogram(xlabel="Countries", ylabel="Distance")函数方法二:get_cluster_labels(num_clusters)
我们把树状图在 k 个簇处切开,给每个实体返回一个整数标签(与 entity_ids 对齐)。一般来说,我们画出树状图就够用了;只有在特定研究需要标签的时候再用这个函数。
用法
# 比如选择 6 个簇
labels = cluster.get_cluster_labels(num_clusters=6)参数(Entry parameters)
| 参数 | 必填 | 类型 | 说明 |
|---|---|---|---|
num_clusters | ✓ | int | 目标簇数 k。内部调用 SciPy 的 fcluster(..., t=k, criterion="maxclust")。 |
返回值(Returns)
- 形状为
(n,)的numpy.ndarray,元素为1...k的整数簇标签。
说明
结果是确定性的(deterministic),只要给同一套距离矩阵和同一种算法,结果通常可以复现。
层次聚类本身不引入随机性(不同于 K-Medoids)。只有在出现完全相等的距离时,可能因为实现细节或行列顺序不同,合并顺序会不一样,树状图形状也会略有区别,但整体的聚类质量基本不受影响。
当 k 很大(≥ n)时,单例(singleton)会很多;当 k=1 时,所有观测都会被放进同一个簇。
单例就是只包含一个实体的簇。k 越接近 n,单例越多,通常可解释性越差;反过来,
k=1则所有实体都在同一簇里。簇标签只是编号,不代表大小或重要程度。
比如 Cluster 1 和 Cluster 2 就是个编号,不代表谁更大、更重要。这里的数字只是用来标识不同的簇。
例子
# 基本用法:假设我们认为 k=6 也就是六个簇比较合适
labels_k6 = cluster.get_cluster_labels(num_clusters=6)
# 合并成整洁表格
import pandas as pd
members = pd.DataFrame({
"Entity ID": cluster.entity_ids,
"Cluster": labels_k6
})
# 多个 k 快速查看规模分布
for k in [3, 4, 5, 6, 8]:
labels = cluster.get_cluster_labels(num_clusters=k)
print(k, pd.Series(labels).value_counts().sort_index().to_dict())错误与警告
如果未生成链接矩阵(Linkage matrix),会报
ValueError: Linkage matrix is not computed.num_clusters需为正整数,否则SciPy的fcluster会报错;请使用 ≥ 1 的整数。
例子
1) 基础流程:拟合、画树、取标签
cluster = Cluster(distance_matrix, ids, clustering_method="ward")
cluster.plot_dendrogram(save_as="dendrogram.png", title="My Dendrogram")
labels_k6 = cluster.get_cluster_labels(num_clusters=6)2) 迷你流水线:质量评估与结果导出
from sequenzo.clustering.hierarchical_clustering import ClusterQuality, ClusterResults
cluster = Cluster(distance_matrix, ids, "ward")
# 用 CQI 比较不同的 k
cq = ClusterQuality(cluster, max_clusters=20)
cq.compute_cluster_quality_scores()
print(cq.get_cqi_table())
cq.plot_cqi_scores(save_as="quality.png")
# 导出 k=6 的结果
cr = ClusterResults(cluster)
members = cr.get_cluster_memberships(num_clusters=6) # 两列:实体 ID 与簇标签
dist = cr.get_cluster_distribution(num_clusters=6) # 统计每个簇的数量与占比
cr.plot_cluster_distribution(num_clusters=6, save_as="cluster_sizes.png")3) 距离矩阵排障措施
# 若矩阵含 NaN/Inf 或轻微不对称,Cluster() 会自动修复。
# 如果你想显式地处理:
D = np.asarray(distance_matrix)
D[~np.isfinite(D)] = np.nanmax(D[np.isfinite(D)])
D = (D + D.T) / 2
cluster = Cluster(D, ids, "average")备注与提示
距离矩阵必须是
n×n,且行列顺序要与entity_ids对齐。如果做了子集或重排,请保持一致。从理论上讲,Ward 更偏好近似平方欧氏距离(approximately squared Euclidean distances)的相异度;实践中,用在很多序列距离上也表现不错,但建议每次都做一下合理性检查(sanity check)。
复杂度:链接计算(computing linkage)在时间与内存上大致为 O(n²)。
这意味着 n 越大,代价按平方增长。例如:
n=1,000:约 100 万对距离,轻松搞定;
n=10,000:约 1 亿对距离,需要几个 GB 内存,可能比较慢;
n=50,000:约 25 亿对距离,一般个人电脑可能难以承受,配置比较高的个人电脑或许能跑下来,但需要比较久的时间。
如果数据非常大(比如超过 50,000 个实体),可以考虑:
推荐使用 CLARA 算法:详见《CLARA 函数文档》 。
去重(Deduplication): 如果在数据集中有大量相同序列,只保留一份并加权,缩小矩阵规模。
抽样(Sampling): 随机抽取一部分实体。
接下来做什么
我们用
ClusterQuality()配合多个 CQI 选择合适的k。我们用
ClusterResults()导出并可视化簇规模,然后:
更多关于 ClusterQuality() 与 ClusterResults() 的细节,请参考接下来的两篇函数文档。
代码:梁彧祺
文档:梁彧祺
编辑:梁彧祺
翻译:明煜坤